羰基还原酶CprCR催化性能提高的定点突变分子改造

羰基还原酶CprCR催化性能提高的定点突变分子改造(任务书,开题报告,论文17000字)
摘要
(S)-N-Boc-3-羟基哌啶((S)-NBHP)作为一种手性醇【1】,是合成多种手性药物的关键中间体,例如非天然药物卡普瑞林,FDA批准抗癌药依鲁替尼(一种Bruton's酪氨酸激酶 (BTK) 抑制剂类的首创新药)【2】。传统的制备(S)- N-Boc-3-羟基哌啶方法一般会采用化学法,这种方法高成本、高能耗、高污染的,限制了其工业应用。而以N-Boc-3-哌啶酮作为潜手性底物的生物酶催化的方法制备(S)-NBHP因其生产成本低,反应条件温和、低污染,逐渐得到研究者重视。然而从目前已有的报道来看,可以催化不对称N-Boc-3-哌啶酮合成(S)-NBHP的酶十分少,由此挖掘具有催化合成(S)-NBHP能力的酶是目前研究的重点。【3】
本次采用基因组数据挖掘的方法进行研究,筛选得到来自于近平滑假丝酵母的羰基还原酶CprCR。将重组质粒pET-22b-CprCR转化至大肠杆菌中表达,酶活测定显示,其粗酶液对底物N-Boc-3-哌啶酮的酶活为5.04U/ml发酵液。将CprCR粗酶液用于催化N-Boc-3-哌啶酮合成(S)-NBHP,催化体系中添加了葡萄糖脱氢酶和辅底物葡萄糖用于辅酶再生【4】。经过24 h的反应, 80g/l的底物最终转化率均能达93%以上,而且产物(S)-NBHP的光学纯度e.e.值>99%,实验数据表明其具有很高的立体选择性与活性。 [资料来源:http://doc163.com]
通过序列比对、同源建模、分子对接等蛋白质工程手段【5】,以CprCR与底物的复合物模型为基础,基于减小空间位阻的原理对影响活性的关键残基55位亮氨酸和285位苯丙氨酸进行定点突变,以期得到双相催化体系中CprCR催化活力提高的酶。结果得到了在水-乙酸丁酯双相催化体系中催化效率更高的突变体L55A,相比野生菌,最终转化率提高了20%,达到了98%。
关键词:(S)-N-Boc-3-羟基哌啶羰基还原酶CprCR双相催化定点突变
Abstract
(S)-N-Boc-3-hydroxypiperidine ((S)-NBHP) is a critical chiral intermediate in the synthesis of pharmaceuticals, including capromorelin and ibrutinib (a Bruton’s tyrosine kinase (BTK) inhibitor), which is the active pharmaceutical ingredient of the new drug Imbruvica approved by FDA for the treatment of lymphoma. The traditional chemical method for producing (S)-N-Boc-3-hydroxypiperidine is high cost, high energy-consumption and high-polluting, which limit the application in the pharmaceutical industry. The bio-enzyme catalysis with N-Boc-3-piperidone as prochiral substrates is a promising method for preparing (S)-NBHP because of its low cost, mild reaction condition and low pollution. However, the enzyme has been reported to catalyze asymmetric reduction to (S)-NBHP is very less. So, discoverying the new enzyme for preparing (S)-NBHP is the main emphasis of the research nowadays.
[资料来源:http://doc163.com]
An (R)-specific carbonyl reductase from Candida parapsilosis was identified for the first time by genome database mining. When recombinant plasmid pET-28a-CprCR with codon optimization expressed in Escherichia coli, recombinant crude enzyme exhibited an activity of 5.04 U/mL fermentation broth with N-Boc-3-piperidone as the substrate. The curde enzyme CprCR was applied to asymmetric bioconversion of N-Boc-3-piperidone to (S)-NBHP, glucose dehydrogenase and cosubstrate glucose were added in catalytic systems for coenzyme regeneration. The result showed that 80 g/L N-Boc-3-piperidone were converted to (S)-NBHP, all of the systems with the conversion of 94% and an e.e. > 99% at 24 h, which means CprCR has high activity and stereo selectivity.
Through sequence alignment, homology modeling, molecular docking and other methods of protein engineering, with the compounds model of CprCR and substrate as the foundation, reduce the space steric hindrance to improve the activity of CprCR in two-phase catalytic system. Site-directed mutagenesis to the key residues 55 leucine and 285 phenylalanine, we obtained a more efficient mutant L55A with the final conversion rate reached 98%, increased by 20%, in water-butyl acetate [资料来源:www.doc163.com]
two-phase catalytic system compared with wild fungus.
KEYWORDS: (S)-N-Boc-3-hydroxypiperidine; Carbonyl reductase CprCR; Cofactor regeneration; Enzymatic properties; Biphase catalysis; site-directed mutagenesis
[来源:http://www.doc163.com]
目 录
摘 要 I
Abstract II
目 录 IV
第一章 文献综述 1
1.1 研究背景 1
1.1.1 羰基还原酶 1
1.1.2生物催化的概念 1
1.1.3 (S)-1-BOC-3-羟基哌啶(S-NBHP)的研究意义及研究前景 1
1.2 (S)-1-BOC-3-羟基哌啶的合成 2
1.2.1 化学制备S-NBHP究进展 2
1.2.2 生物制备S-NBHP研究进展 2
1.3酶分子改造方法的研究进展 3
1.3.1理性设计方法 3
1.3.2定向进化技术 3
1.3.3半理性设计方法 4
1.3.4催化活性改造 4 [资料来源:www.doc163.com]
1.4 羰基还原酶定点突变分子改造 4
1.4.1 羰基还原酶概述 4
1.4.2 羰基还原酶的定点突变理性设计 4
1.4.3 羰基还原酶催化活性改造 5
1.5 本论文的研究意义和主要研究内容 5
1.5.1 立题背景和意义 5
1.5.2 本论文的主要研究内容 6
第二章 羰基还原酶CprCR的克隆表达及功能鉴定 7
2.1 前言 7
2.2 材料和方法 7
2.2.1 实验材料 7
2.2.2 实验试剂 8
2.2.3 工具酶及试剂盒 9
2.2.4 菌株与质粒 10
2.2.5 培养基及培养方法 11
2.2.5.1 培养基 11
2.2.5.2 菌株培养方法 11
2.3 实验方法 12
2.3.1 近平滑假丝酵母基因组的提取 12 [资料来源:https://www.doc163.com]
2.3.2 重组质粒pET-22b-CprCR的构建及鉴定 13
2.3.3 目的基因CprCR的密码子优化 14
2.3.4 目的蛋白CprCR的表达 14
2.3.5 底物及产物的检测方法 14
2.3.5.1 N-Boc-3-哌啶酮与NBPH的测定方法 14
2.3.5.2 N-Boc-3-哌啶酮与(S)-NBPH、(R)-NBPH手性鉴定方法 15
2.3.6 双酶偶联催化不对称还原N-Boc-3-哌啶酮 15
2.4 结果与讨论 15
2.4.1 基因组数据挖掘获得羰基还原酶CprCR 16
2.4.2 近平滑假丝酵母基因组的提取 16
2.4.3 目的基因的PCR扩增 17
2.4.4 重组质粒的构建和PCR鉴定 17
2.4.5 CprCR的蛋白表达 18
2.4.6 CprCR的酶活测定 18
2.4.7 N-Boc-3-哌啶酮与NBPH的测定及标准曲线的绘制 19
2.4.8 N-Boc-3-哌啶酮与(S)-NBPH、(R)-NBPH手性鉴定方法 20 [资料来源:http://www.doc163.com]
2.4.9 双酶偶联催化不对称合成(S)-NBHP初探 20
2.5 本章小结 21
第三章 羰基还原酶CprCR的定点突变提高催化活性 22
3.1 前言 22
3.2 实验材料与方法 23
3.2.1 实验仪器 23
3.2.2 实验试剂 23
3.2.3 工具酶 23
3.2.4 菌株与质 23
3.2.5 培养基与培养方法 23
3.3 实验方法 23
3.3.1 序列比对、同源建模及结构分析 23
3.3.2 突变引物设计 23
3.3.3 定点突变的方法 24
3.3.4 突变菌株的初步筛选与鉴定 25
3.3.4.1 变菌的酶活测定 25
3.3.4.2 变菌的蛋白表达 25
3.3.5 突变菌株催化合成(S)-NBHP反应 25
3.3.5.1 底物浓度为50 g/l的催化 25 [来源:http://Doc163.com]
3.3.6 利用最优突变菌构建双酶共表达重组质粒 25
3.3.7 最优变菌株催化高底物浓度的反应研究 25
3.3.8 突变菌株的纯化及动力学参数测定 26
3.4 结果与讨论 26
3.4.1 序列比对、分子对接结果 26
3.4.2 突变残基的选择 26
3.4.3 突变酶的酶活测定与蛋白表达 27
3.4.3.1 酶活测定 27
3.4.3.2 蛋白表达 28
3.4.4 底物浓度为50 g/l筛选的突变菌株催化合成(S)-NBHP 28
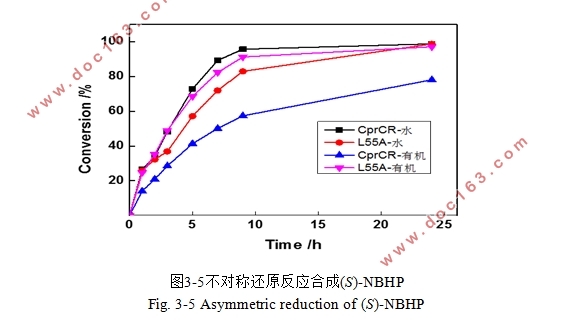
3.4.5 最优突变菌株共表达催化高底物浓度的反应 29
3.4.6 突变酶的纯化及动力学参数比较 30
3.5 本章小结 31
第四章 结论与展望 31
4.1 结论 31
4.2 展望 32
参考文献 32
致 谢 35
